AnalyticDB for MySQL是雲端託管的PB級高併發低延時數據倉庫,通過AnalyticDB for MySQL向量檢索功能構建基因檢索系統,支持毫秒級針對10億級別的向量數據進行查詢分析,更加快速、高效地為新冠肺炎病毒防控、研發治療藥物以及相關疫苗提供幫助。
1.背景信息
2019年年底,中國的新興商業中心武漢,爆發了一種名為新型冠狀病毒肺炎(簡稱新冠肺炎)的疫情。在新冠肺炎流行的兩個多月中,中國造成了3300多人死亡,8萬2千多人感染。隨著疫情的進一步蔓延,目前已經橫跨了109個國家,造成了80多萬人感染,4萬多人失去了生命。到目前為止,疫情使得50多個國家停擺,全世界範圍內造成了數千億美元的經濟損失。在疫情防治期間,阿里雲提供了高效基因序列檢索技術,助力新冠肺炎病毒序列分析。
2.基因序列檢索技術應用範圍和現狀
對於當下疫情,基因序列檢索技術主要應用於以下場景:
- 用於新冠肺炎的溯源和分析,幫助疫情防控找到病毒宿主,做好有效防範。
通過基因序列檢索技術,發現蝙蝠和穿山甲身上的冠狀病毒RNA序列匹配度高達96%和99.7%,可以推斷蝙蝠和穿山甲很可能是新冠肺炎的宿主。 - 用於分析新冠肺炎病毒的複製和傳播過程,為研發治療藥物和疫苗提供幫助。
通過基因序列檢索技術對基因序列按功能區域劃分,瞭解各個模塊的功能,從而更好地分析出病毒的複製和傳播過程,進而找到關鍵節點,為研發治療藥物和疫苗提供幫助。 - 用於檢索到與冠狀病毒相似的病毒基因序列。
基因序列檢索技術也可以檢索與新冠肺炎病毒相似的病毒基因序列,例如SARS、MERS等病毒,從而借鑑相關藥物靶點設計機制,更快、更高效地研發檢測試劑盒、疫苗以及相關的治療藥物。
基於當下疫情的快速蔓延,當前的基因匹配算法太慢,迫切需要高效匹配算法進行基因序列檢索。阿里雲AnalyticDB for MySQL團隊將基因序列片段轉化成對應的1024維特徵向量,將兩個基因片段的匹配問題,轉換成了兩個向量間的距離計算問題,從而大大降低了計算開銷,實現毫秒級返回相關基因片段,完成基因片段的首次篩選。然後,使用基因相似計算BLAST算法,完成基因相似度的精確排查,從而高效率完成基因序列的匹配計算。匹配算法從原來O(M+N)的複雜度降低到O(1)。同時,阿里雲AnalyticDB for MySQL提供強大的機器學習分析工具,通過基因轉向量技術,將局部的和疾病相關的關鍵靶點基因片段轉成特徵向量,用於基因藥物的研發,大大加速了基因分析過程。
3. AnalyticDB for MySQL基因檢索系統
新冠肺炎病毒的RNA序列可以用一串核酸序列(又稱鹼基序列)表示,RNA序列含有四種核苷酸,分別用A、C、G和T表示,分別代表腺嘌呤、胞嘧啶、鳥嘌呤、胸腺嘧啶。每個字母代表一種鹼基,無間隔排列在一起。每一個物種的RNA序列均不相同但又有規律,基因檢索系統可以通過輸入一串病毒的基因片段,檢索相似的基因,用來對病毒的RNA序列進行分析。
為方便演示AnalyticDB for MySQL基因片段檢索方法,我們從GenBank下載了大量病毒的RNA片段,並將GenBank內部關於病毒的論文以及Google Scholar中相關病毒的論文導入AnalyticDB for MySQL基因檢索數據庫中。
AnalyticDB for MySQL基因檢索的演示界面如下圖所示,將新冠肺炎病毒的序列(COVID-19)上傳到AnalyticDB for MySQL基因檢索系統中,AnalyticDB for MySQL基因檢索系統只需幾毫秒即可檢索到相似的基因片段(當前示例系統只返回匹配度超過0.8的基因片段)。從返回的基因片段得出穿山甲攜帶的新冠肺炎病毒(GD/P1L)、蝙蝠攜帶的新冠肺炎病毒(RaTG13)以及SARS和MARS病毒,其中GD/P1L的序列匹配度最高為0.974,由此推斷出新冠肺炎病毒很可能是通過穿山甲傳染到人的。
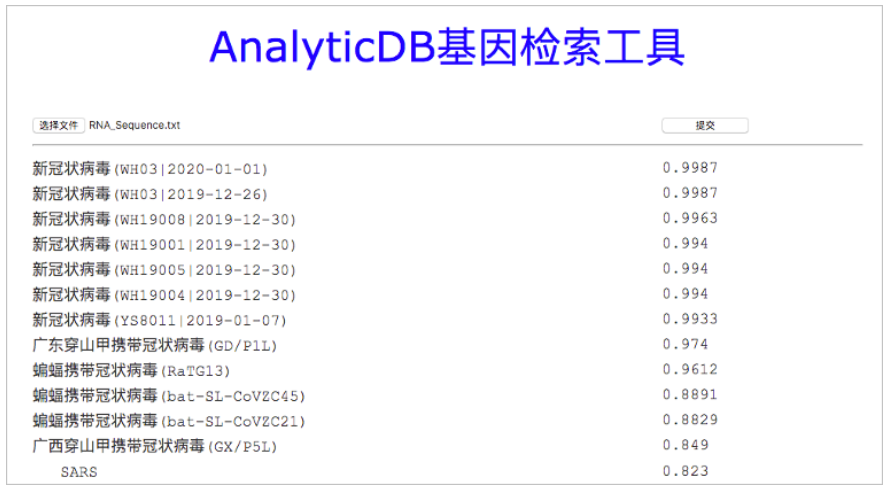
圖1:AnalyticDB for MySQL基因檢索演示界面
如果RNA片段非常相似,說明這兩個RNA可能有相似的蛋白質表達和結構。通過基因檢索工具,可以看到SARS和MARS與新冠肺炎病毒的匹配度為0.8以上,說明可以將一些SARS或者MARS的研究成果應用到新冠肺炎病毒上。系統提取了每種病毒的論文,通過文本分類算法,將論文劃分為檢測類、疫苗類和藥物類,例如下圖為SARS的七種檢測方法、四種疫苗接種方法和10中治療藥物。其中,對SARS有效的熒光定量PCR檢測,目前正應用於新冠肺炎病毒的檢測;基因疫苗的方法以及誘導體內免疫疫苗的方法,也正在展開研究;治療藥物中瑞德西韋以及相關的干擾素也都用於新冠肺炎病毒的治療上。

圖2:SARS病毒相關論文檢索分類結果
單擊上圖干擾素鏈接可以查看相關論文,當前系統調用了自動翻譯軟件,抽取中文版文件名關鍵詞作為文件名,方便用戶閱讀。
圖3:論文查看效果
4.實現架構
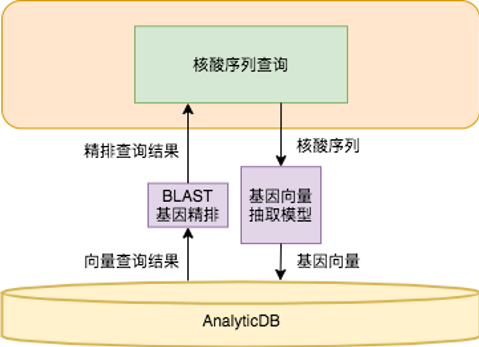
圖4:基因檢索實現架構
AnalyticDB for MySQL基因檢索系統中,AnalyticDB for MySQL負責存儲和查詢所有結構化數據(例如基因序列的長度,長度包含基因的論文名稱、基因種類、DNA或者RNA等)和基因序列產生的特徵向量。查詢數據時,通過基因向量抽取模型將基因轉化成向量,在AnalyticDB for MySQL向量數據庫中進行粗排檢索,然後在返回的向量匹配結果集中使用經典的BLAST算法進行精確檢索,返回最相似的基因序列。
AnalyticDB for MySQL基因檢索系統的核心是基因向量抽取模型,該模塊可以將核苷酸序列轉化成向量。目前AnalyticDB for MySQL抽取了各種病毒的RNA全部序列樣本進行訓練,可以非常方便地對病毒的RNA進行相似度計算。同時,基因向量抽取模型也可以擴展應用於其他物種基因檢索。
5.基因向量抽取算法
目前詞向量技術已經非常成熟,被廣泛應用於機器翻譯、閱讀理解、語義分析等相關領域,並取得了巨大成功。詞向量化採用了分佈式語義的方法表示一個詞的含義,一個詞的含義就是這個詞所處的上下文語境。例如高中英語中的完形填空題,一篇短文空出10個地方,學生根據空缺詞的上下文語境選擇合適的詞。也就是說上下文語境能夠準確地表達這個詞,如果某位同學選擇了正確的選詞,表示該同學理解了空缺詞的含義。因此,通過上下文詞的關係,採用詞向量算法,可以為每個詞生成一個向量,通過計算兩個詞向量之間的相似度,得到兩個詞的相似度。
同樣的道理,基因序列的排列具有一定的規律,並且每一部分基因序列所表達的功能和含義不同。可以將很長的基因序列劃分成小的單元片段(也就是詞)進行分析,並且這些詞也有上下文語境,這些詞相互連接、相互作用共同完成相對應的功能,形成合理的表達。因此,生物科學家們採用詞向量算法對基因序列單元進行向量化,兩個基因單元相似度很高,說明需要這兩個基因單元共同來表達和完成相應的功能。
總體而言,AnalyticDB for MySQL基因向量抽取算法分為三步:
(1)在氨基酸序列中定義詞。生物信息學中用K-mers來分析氨基酸序列,K-mer是指將核酸序列分成包含K個鹼基的字符串,即從一段連續的核酸序列中迭代選取長度為K個鹼基的序列,若核酸序列長度為L,K-mer長度為K,那麼可以得到L-K+1個K-mers。例如下圖所示,假設某序列長度為12,設定選取的k-mer長度為8,則得到(12-8+1=5)5個5-mers。這些k-mer,就是氨基酸序列中的詞。
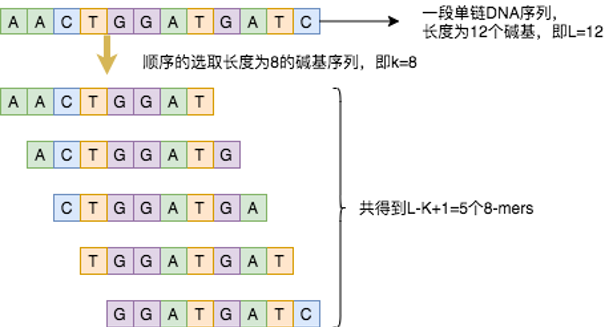
圖5:8-mers核酸序列示意圖
(2)找到氨基酸序列的上下文語境,將基因序列中的詞轉換成1024維向量。對於詞向量算法而言,另一個重要的問題就是上下文的語境。AnalyticDB for MySQL基因向量抽取算法在氨基酸片段中選擇一個長度為L的窗口,該窗口內的氨基酸片段可認定為在同一語境內。例如CTGGATGA是一段核酸序列,選取了長度為10的窗口,AnalyticDB for MySQL基因向量抽取算法將CTGGATGA轉換成5個5-mers即{AACTG, ACTGG, CTGGA, GGATG, GATGA}。對於其中一個5-mer{CTGGA}而言,另外四個{AACTG, ACTGG, GGATG, GATGA}5-mers就是當前5-mer {CTGGA}的上下文語境。AnalyticDB for MySQL基因向量抽取算法套用詞向量空間訓練模型,對已有生物基因的K-mers進行訓練,便可將一個K-mer(基因序列中的一個詞)轉換成1024維向量。
(3)類似於詞向量模型,K-mer向量模型也擁有和詞向量模型相似的數理計算性質。公式一說明核苷酸序列ACGAT的向量減去GAT序列的向量和AC序列的向量距離非常接近。公式二說明核苷酸序列AC的向量加上ATC序列的向量和ACATC序列的向量距離也很接近。因此,根據這些數理特徵,計算一個長氨基酸序列向量時,可以將這個序列中每一段的K-mer序列進行累加,最後進行歸一化就能得到整個氨基酸序列的向量。同時,為提升精度,可以將基因片段看作一個文本,使用doc2vec4將整個序列轉換成向量進行計算。為進一步驗證算法性能,AnalyticDB for MySQL基因向量抽取算法計算了常用於基因檢索庫中的BLAST算法序列與基因轉向量l2距離序列的相似度,兩個序列的斯皮爾曼等級相關係數是0.839。以上得出結論,將DNA序列轉換成向量用於相似基因片段的初次篩選,是有效且可行的。
6.向量檢索功能概述
一般包含向量檢索的應用系統中,開發者通常會使用向量檢索引擎(例如Faiss)存儲向量數據,然後使用關係型數據庫存儲結構化數據。因此,查詢時也需要交替查詢兩個系統,明顯額外增加了開發人員的工作量,數據查詢性能也不是最優。
AnalyticDB for MySQL是雲端託管的PB級高併發低延時數據倉庫,可以毫秒級針對10億級別的向量數據進行查詢,100毫秒級別的響應時間。AnalyticDB for MySQL全面兼容MySQL協議以及SQL:2003語法標準,其向量檢索功能支持對圖像、文本推薦、聲紋、核苷酸序列等相似性進行查詢和分析,目前在多個城市的安防項目中已大規模部署了AnalyticDB for MySQL。
AnalyticDB for MySQL支持結構化和非結構化數據的近似檢索和分析,通過SQL接口即可快速搭建基因檢索或者基因+結構化數據混合檢索等系統。在混合檢索場景中AnalyticDB for MySQL的優化器會根據數據的分佈和查詢條件選擇最優執行計劃,在保證數據召回率的同時,得到最優的性能。例如,通過以下一條SQL即可檢索RNA核酸序列。
-- 查找RNA和提交的序列向量相近的基因序列。
select title, # 文章名
length, # 基因長度
type, # mRNA或DNA等
l2_distance(feature, array[-0.017,-0.032,...]::real[]) as distance # 向量距離
from demo.paper a, demo.dna_feature b
where a.id = b.id
order by distance; # 用向量相似度排序
上述SQL中表demo.paper用於存儲上傳的每篇文章的基本信息,demo.dna_feature存儲各個物種的基因序列對應的向量。通過基因轉向量模型,將要檢索的基因轉成向量[-0.017,-0.032,...],然後在AnalyticDB for MySQL數據庫中進行檢索。
當前系統也支持結構化信息+非結構化信息(核苷酸序列)的混合檢索,例如查找和冠狀病毒相關的類似基因片段時,只需要在SQL中增加where title like'%COVID-19%'即可。
本文參考的相關文獻,感興趣的讀者可訪問這裡或掃描下方二維碼瞭解詳情。

作者:李倩
阿里云云原生數據倉庫AnalyticDB產品經理
阿里云云原生數據倉庫AnalyticDB產品經理,見證了AnalyticDB從初創到至今的壯大發展,參與了所有重大功能設計。
我們是阿里雲智能全球技術服務-SRE團隊,我們致力成為一個以技術為基礎、面向服務、保障業務系統高可用的工程師團隊;提供專業、體系化的SRE服務,幫助廣大客戶更好地使用雲、基於雲構建更加穩定可靠的業務系統,提升業務穩定性。我們期望能夠分享更多幫助企業客戶上雲、用好雲,讓客戶雲上業務運行更加穩定可靠的技術,您可用釘釘掃描下方二維碼,加入阿里雲SRE技術學院釘釘圈子,和更多雲上人交流關於雲平臺的那些事。
